機械学習に基づくタンパク質の機能予測
機械学習を用いて、タンパク質の機能を予測する手法の開発に取り組んでいます。
生合成遺伝子機能予測データベースの構築
米国生物工学情報センター(National Center for Biotechnology Information; NCBI)が管理している非冗長配列データベースReference Sequence Database(RefSeq)には、3億を超えるタンパク質のアミノ酸配列が登録されています。一方、欧州バイオインフォマティクス研究所(European Bioinformatics Institute; EBI)が運営するタンパク質データベースUniProt Knowledgebase(UniProtKB)に登録されているタンパク質のうち、専門家によって機能が確認され、SwissProtに登録されているタンパク質は57万に過ぎません。本研究では、配列データベースに埋もれている多数の機能未知タンパク質の機能を予測し、有用な反応を触媒する酵素を見出すことを目指しています。
私たちは予測結果を登録したデータベースを構築し、Webサーバ(http://sr.iu.a.u-tokyo.ac.jp/)にて公開しています(図1)。ここでは、配列類似性に基づく機能予測結果に加えて、AlphaFoldによる予測構造を閲覧できるようになっています。今後は、現在開発中の機械学習による機能予測結果を加え、さらに使いやすいデータベースに拡充することを目指しています。
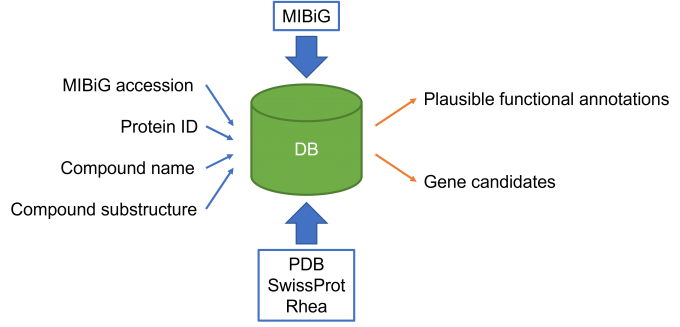
図1: データベースの概要。生合成遺伝子クラスタデータベースMIBiGに登録されたタンパク質について、PDB、SwissProtに登録されているタンパク質との配列相同性を行い、結果をデータベース化している。また、酵素反応データベースRheaと連携して、触媒する酵素反応のデータも登録している。Webサーバでは、MIBiG accessionの他、Protein IDや反応に参加する化合物名や化合物の部分構造を用いて、タンパク質を検索し、その予測された機能を取得することができる。
タンパク質の機能同一性予測法の開発
多数の機能未知タンパク質から有用な反応を触媒する新規酵素を効率的に見出すためには、その基質と生成物、すなわち機能を、コンピューターを用いて予測する方法が必要となります。よく似たアミノ酸配列を持つ酵素は、同じ機能を持つ傾向があることから、BLAST検索がよく用いられますが、その能力は十分とは言えません(図2参照)。そこで私たちは、アミノ酸配列の類似性に加えて、立体構造の類似性を用いて、2つの酵素が同じ機能を持つかどうかを予測する方法を開発しました。私たちはこの方法を、FUJISAN(FUnctional identity of protein prediction by JoIning Sequence ANd structure feature)と名付けました。図2は、FUJISANの予測性能の、配列類似性(E-value)のみに基づく方法、および深層学習(ESM-2およびDeepFRI)に基づく方法の予測性能との比較を表しています。FUJISANはこれらの方法よりも優れた予測性能を示しました。FUJISANのコードはGithubにて公開しています。現在私たちは、FUJISANを用いてMIBiGに登録された生合成酵素の網羅的な機能予測に取り組んでいます。
Fujita and Terada, Comput. Struct. Biotechnol. J. 23, 4124–4130 (2024).
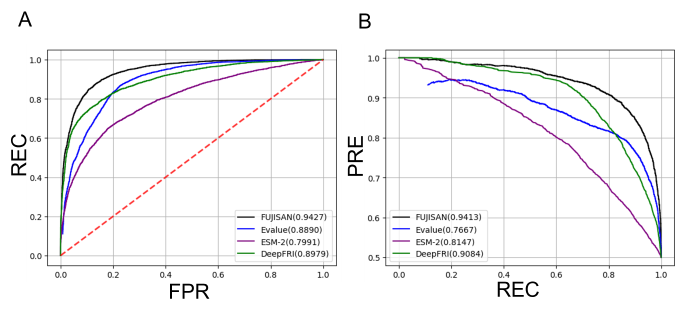
図2: FUJISANと配列類似性(E-value)のみに基づく方法、ESM-2に基づく方法、DeepFRIに基づく方法のROC(receiver operating characteristic)曲線(A)と適合率-再現率曲線(B)。REC、FPR、PREはそれぞれ、再現率、偽陽性率、適合率を表す。ROC曲線および適合率-再現率曲線の下の部分の面積の値をカッコ内に示す。この値が大きいほど、予測性能が高いことを表している。
