粗視化MD法の開発と応用
細胞スケールのシミュレーションを目指して、粗視化MD法の開発と応用を行っています。
リガンド結合過程の粗視化MDシミュレーション
リガンドがタンパク質に結合する過程(図1)の時間スケールはミリ秒程度の遅い過程であるため、すべての原子を考慮した全原子MD法を用いてこの過程を追跡することは困難です。また、リガンド結合過程は確率過程であるため、多数回の試行(シミュレーション)が必要となります。本研究では、粗視化MD法によりリガンド結合過程を追跡する方法の開発を行いました。
ここでは、粗視化モデルとしてMARTINIを使用しました。このモデルでは、平均4つの非水素原子を1つの粗視化粒子にマップします。相互作用が異なる18種類の粒子種が用意され、アミノ酸の粗視化モデルには、油水分配係数を再現するように、粒子種が割り当てられています。ここでは、タンパク質としてlevansucrase、リガンドとしてsucroseを選定し、粗視化MDシミュレーションを行いました。タンパク質の周囲にリガンドをランダムに配置し、4 μsのMDシミュレーションを、初速とリガンドの初期配置を変えて50回繰り返しました。リガンドが基質ポケットに結合する様子を図2に示します。このシミュレーションにより、49回の結合と18回の解離を観測しました。この結果から、結合・解離速度定数と、解離定数を計算したところ、解離定数は実験値とほぼ一致し、結合・解離速度定数は実験値の10倍程度となりました。粗視化MDシミュレーションでは、分子間の摩擦が小さいため、分子は速く拡散することが知られています。これを考慮すると、粗視化MDシミュレーションにより、タンパク質へのリガンド結合過程のダイナミクスがよく再現されたと言えます。さらに私たちは、リガンドが基質ポケットに近づいている時間のトラジェクトリを用いてリガンドの流速を計算しました。この結果、リガンドは特定の経路を通って基質ポケットに結合することと、リガンド結合経路は、タンパク質表面上の溝に沿う傾向があることが示されました。
Negami et al., J. Comput. Chem. 35, 1835 (2014).
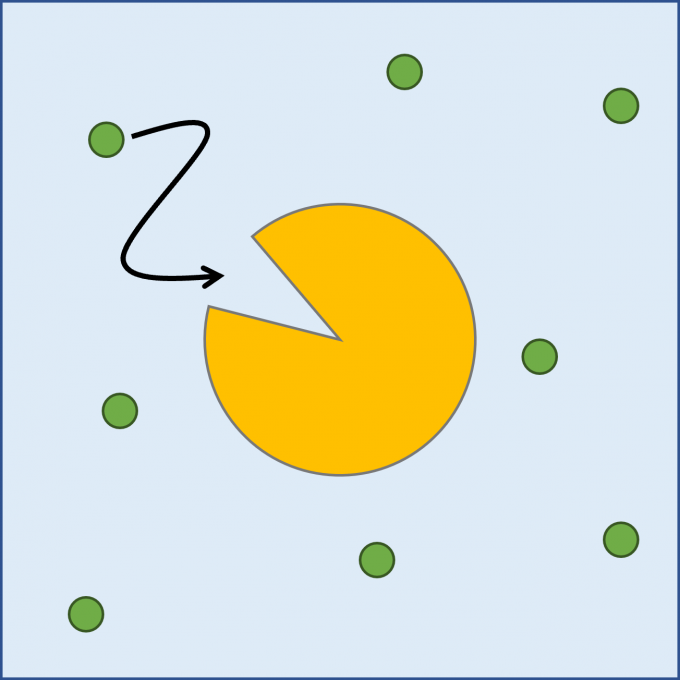
図1: リガンド結合過程の模式図。中心の切れ込みのある黄色い物体がタンパク質、周囲の緑色の球がリガンド、水色の地が水溶液を示している。リガンドは、切れ込みで表したタンパク質の基質ポケットに向けて運動し、結合する。
図2: 粗視化MDシミュレーションで観察された、リガンドの基質ポケットへの結合の様子。リガンドをスティックモデルで示した。タンパク質の表面のうち、親水性アミノ酸による表面を水色、疎水性アミノ酸による表面をオレンジ色で示した。
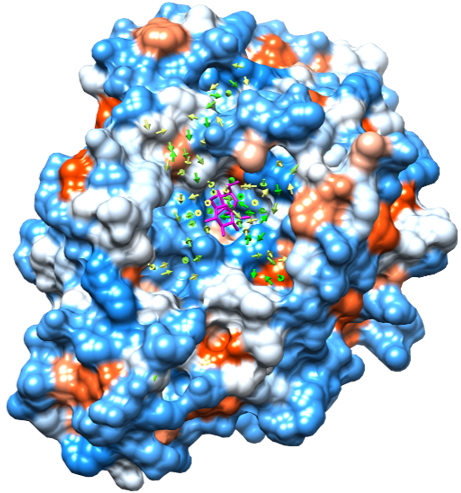
図3: リガンドの流速。タンパク質の表面の色分けは図2と同じ。リガンドの流速を流速の大きさに従って、黄色から緑色の矢印で 示した。結晶構造におけるリガンドの結合構造を紫色のスティックモデルで示した。
リガンド結合に共役して立体構造変化するタンパク質粗視化モデルの開発
タンパク質の中には、リガンドとの結合に伴って立体構造変化するものがあることが知られています。本研究では、タンパク質の粗視化モデルに、立体構造変化前後の2つの状態をエネルギー極小状態とする弾性ネットワークモデルを組み合わせることで、リガンド結合に共役して立体構造変化するタンパク質粗視化モデルを開発しました。リガンド結合に共役した立体構造変化には、リガンド非結合状態で、2つの構造の間の平衡状態にあり、一方の構造のみに結合する”conformational selection”と、リガンド非結合状態のタンパク質にリガンドが結合した後、構造が変化する”induced fit”の2つの過程があります。本研究で開発したモデルでは、パラメータを調節することで、この両方の過程を再現できることを示しました。
Negami et al., Chem. Phys. Lett. 742, 137144 (2020).
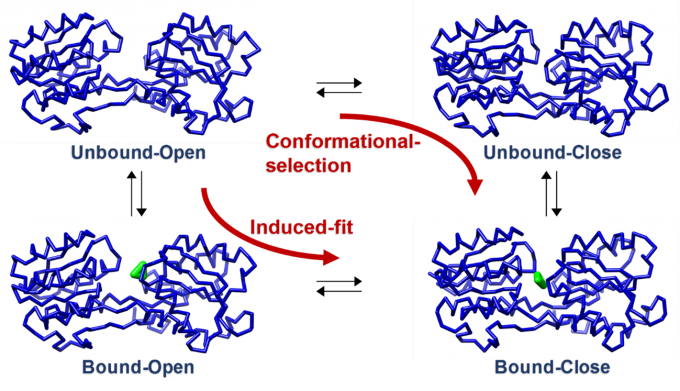
図4: リガンド結合に共役して立体構造変化するタンパク質粗視化モデル。タンパク質を青色のモデルで、リガンドを緑色のモデルで示した。このタンパク質は、リガンド非結合状態では開いた構造を、リガンド結合状態では閉じた構造をとる。
