量子化学計算を用いた酵素の触媒機構の解析
量子化学計算を用いて、酵素が触媒する反応のメカニズムを明らかにする研究を行っています。
メチル基転移酵素Fur6の触媒機構の解析
抗腫瘍活性を持つ化合物furaquinocin Dの生合成経路で機能するメチル基転移酵素Fur6は、S-adenosylmethionine(SAM)から1,2,4,5,7-pentahydroxynaphtalene(PHN)の3位にメチル基を転移する反応を触媒することが知られています(図1)。本研究では、Fur6と、SAMがメチル基を供与した後の生成物であるS-adenosyl-l-homocysteine(SAH)が結合した結晶構造を用いて、Fur6・SAM・PHNの3者複合体の立体構造のモデリングを行い、MDシミュレーションを用いて複合体モデルの安定性を確認した後、QM/MM法を用いて触媒機構解析を行いました(図2)。この結果、活性化エネルギーは8.6 kcal/molであることが示され、反応が常温で進行するという実験結果と一致する結果が得られました。本研究は、応用生命工学専攻の分子育種学研究室と共同で行いました。
Zhao et al., Biochemistry, 63, 806–814 (2024).

図1: 基質PHN(左)と生成物(右)の構造。
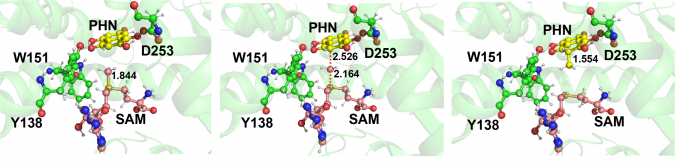
図2: 反応系(左)、遷移状態(中)、中間状態(右)の構造。反応系に対する遷移状態、中間状態のエネルギーは、それぞれ8.6 kcal/mol、−32.9 kcal/mol。中間状態には3位にプロトンが結合しており、水溶液中に放出された後、脱プロトン化されて生成物となる。
ストリゴラクトン合成酵素の立体選択性制御機構の解析
植物ホルモンの一種であるストリゴラクトンは、4つの環構造(A環、B環、C環、D環)を持っていますが、合成の最終段階では、A環、D環のみを持つ前駆体の18-oxo-carlactonoic acid(18-oxo-CLA)からB環、C環が形成します(図3)。この反応をフラスコ内で行うと、環の立体配置が異なるストリゴール型とオロバンコール型が、それぞれ56%、44%生成することが知られています。神戸大学の杉本教授らのグループでは、トマトからこの反応を触媒する酵素SlSRFを同定しました。SlSRFはオロバンコール型のみを生成することから、この酵素の立体選択性制御機構を明らかにすることを目指しました。
本研究ではまず、AlphaFoldを用いてストリゴラクトン合成酵素の立体構造を予測し、基質との複合体のモデリングを行いました。続いて、このモデルに対してMDシミュレーションを行ったところ、酵素内で基質はオロバンコール型の生成に有利なコンフォメーションに固定されていることが示されました。一方、基質のみを水溶液中の環境でMDシミュレーションを行うと、ストリゴール型の生成に有利なコンフォメーションとオロバンコール型の生成に有利なコンフォメーションの両方が現れました(図4)。これは、フラスコ内の反応では両方の型が生成されるという実験事実と一致していることから、反応前の基質のコンフォメーションによって生成物の立体配置が決まることがわかりました。QM/MM法を用いて触媒機構の解析を行った結果、遷移状態1-中間状態-遷移状態2を経由して触媒反応が進行することが明らかとなりました。この過程の活性化エネルギーの最大値は14.6 kcal/molとなり、反応が常温で進行するという実験結果と一致する結果が得られました(図5)。本研究は、神戸大学の杉本教授らのグループと共同で行いました。
Homma et al., Proc. Natl. Acad. Sci. USA, 121, e2313683121 (2024).

図3: ストリゴラクトン合成反応。前駆体の18-oxo-CLAのプロトン化によって反応が進行する。
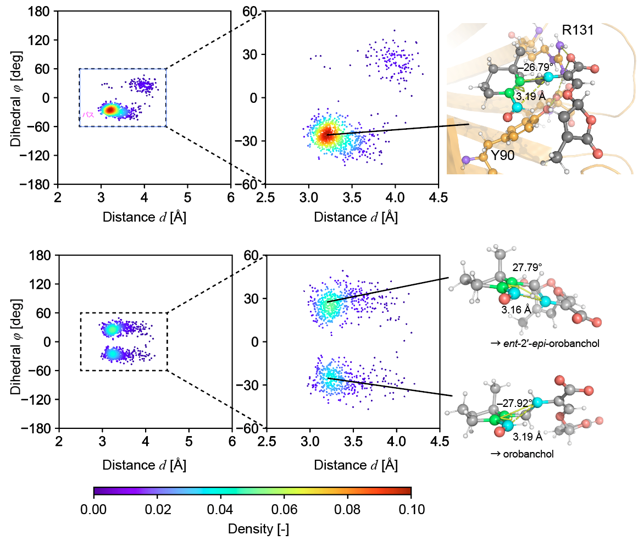
図4: MDシミュレーションにおける18-oxo-CLAのコンフォメーション分布。酵素との複合体(上段)ではオロバンコール型の生成に有利なコンフォメーションが優勢であるのに対し、水溶液中(下段)では両方の方が生成可能なコンフォメーションが現れている。
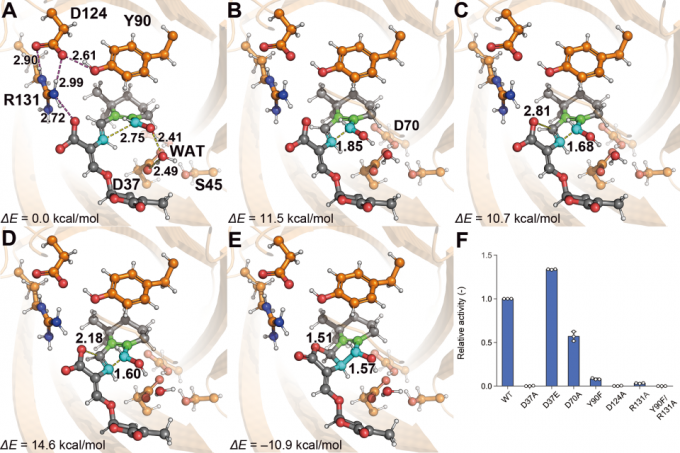
図5: (A)反応系、(B)遷移状態1、(C)中間状態、(D)遷移状態2、(E)生成系の構造、(F)変異体実験による複合体モデルの検証結果。
