
|
�P�D�P ��`�q�\���c�[���Ƃ́H |
��
��`�q�\���c�[���ɂ��Ă̊�{�I�Ȑ����ł��B
�� ��`�q�\���c�[���̋@�\�B
�قƂ�ǂ̈�`�q�\���c�[���́A�Q�m���̔z��f�В��ɑ��݂���G�L�\�������������`�q�����v���O�����ł��BDNA�z�����͂���ƁA���̔z��ɃR�[�h����Ă����`�q�\����\�����A�o�͂��܂��B
�� ��`�q�\���c�[���̗��p�ɂ�������
�w�p�I�ȗ��p�ɂ͖����Ŕz�z����Ă�����̂������A�R���s���[�^��ɃC���X�g�[�����邱�Ƃ��ł��܂����A��ʓI�ɂ͑�w�⌤���������Ă���T�[�o�ɃA�N�Z�X���ė��p���܂��B
|
�P�D�Q ��`�q�\���c�[���̗��p�ɕK�v�ȃR���s���[�^�� |
����`�q�\���c�[���𗘗p����O�ɁA�K�v�Ȋ����m�F���܂��傤�B
�� �u���E�U�i�C���^�[�l�b�g�G�N�X�v���[���[�A�l�b�g�X�P�[�v�i�r�Q�[�^�[�Ȃǁj
�u���E�U���K�v�ł��B��`�q�\���c�[���̃z�[���y�[�W�ɃA�N�Z�X���Ĉ�`�q�\���c�[���𗘗p���܂��B
��
�C���^�[�l�b�g�ڑ�
�C���^�[�l�b�g���g�p�\�ȏ�ԂɂȂ��Ă��邱�Ƃ��O��ł��B�܂��A���[���A�h���X�����L���Ă���A�\�����ʂ�E���[���Ŕz�M���Ă��炤���Ƃ��ł���c�[��������܂��B
|
�P�D�R ���낢��Ȉ�`�q�\���c�[�� |
��
��`�q�\���c�[�����Љ�܂��B
GENSCAN
|
|
|
�ΏۂƂ��鐶���� |
��
�Ғœ����iVertebrate�j ��
�g�E�����R�V�iMaize�j ��
�V���C�k�i�Y�i�iArabidopsis�j |
|
�o�[�W���� |
1.0 �i2001�N6�����݁j |
|
���� |
DNA�z��i�����F���100���i1Mbp�j�j |
|
���͌`�� |
��
HTML�t�H�[�� ��
�e�L�X�g�t�@�C�� ��
E���[�� |
|
�o�͌`�� |
��
�u���E�U��̏o�� ��
�摜�iPDF�A�|�X�g�X�N���v�g�j ��
E���[�� |
|
�g�p�A���S���Y�� |
�B��}���R�t���f�� |
|
�T�[�r�X�������T�[�o |
��
�}�T�`���[�Z�b�c�B�H�ȑ�w�i�A�����J�jhttp://genes.mit.edu/GENSCAN.html ��
�p�X�c�[���������i�t�����X�jhttp://bioweb.pasteur.fr/seqanal/interfaces/genscan.html |
GRAIL
|
|
|
�ΏۂƂ��鐶���� |
��
�q�g�ihuman�j ��
�}�E�X�imouse�j ��
�V���C�k�i�Y�i�iArabidopsis�j ��
Drosophila�i�L�C���V���E�W���E�o�G���j ��
E.coli�i�咰�ہj |
|
�o�[�W���� |
Version1.3 �i2001�N6�����݁j |
|
���� |
DNA�z��i�����F���20���i200kbp�j�j |
|
���͌`�� |
��
HTML�t�H�[�� ��
�e�L�X�g�t�@�C�� |
|
�o�͌`�� |
��
�u���E�U��̏o�� ��
�e�L�X�g�t�@�C�� |
|
�g�p�A���S���Y�� |
�j���[�����l�b�g |
|
�T�[�r�X�������T�[�o |
��
�I�[�N���b�W�����������i�A�����J�jhttp://compbio.ornl.gov/Grail-1.3/ ��
������w��Ȋw�������i���{�jhttp://grail.genome.ad.jp/ |
GeneMark
|
|
|
�ΏۂƂ��鐶���� |
��
�咰�ہiE.coli�j ��
�͑��ہiS.cerevisiae�j ��
T4�t�@�[�W�iPhage_T4�j ��
�q�g�iH.sapiens�j �ȂǑ����̐�����ɑΉ����� |
|
�o�[�W���� |
Version2.4 �i2001�N6�����݁j |
|
���� |
DNA�z��i�����F���100���i1Mbp�j�j |
|
���͌`�� |
��
HTML�t�H�[�� ��
�e�L�X�g�t�@�C�� |
|
�o�͌`�� |
��
�u���E�U��̏o�� |
|
�g�p�A���S���Y�� |
�B��}���R�t���f�� |
|
�T�[�r�X�������T�[�o |
��
�W���[�W�A�H�ȑ�w�i�A�����J�jhttp://genemark.biology.gatech.edu/GeneMark/ ��
���B���q�����w�������i�t�����X�jhttp://www.ebi.ac.uk/genemark/ |

|
�Q�D�P GENSCAN�̃z�[���y�[�W�Ɉړ� |
�� �܂��A�C���^�[�l�b�g�G�N�X�v���[���[�iInternet Explorer�j���g����GENSCAN�ɃA�N�Z�X���܂��傤�B
�@ �m�X�^�[�g�n�|�m�v���O�����n�|�mInternet Explorer�n���N���b�N����B
�A
�A�h���X�̓��͗���GENSCAN����Ă���T�C�g��URL
http://genes.mit.edu/GENSCAN.html
����͂��܂��B
�B Enter�L�[�������ƁAGENSCAN�̃z�[���y�[�W���\������܂��B

��`�q�\���c�[���������z�[���y�[�W
|
�Q�D�Q DNA�z��̓��� |
�� ��`�q�\���\�����s��DNA�z�����͂��܂��傤�B
�@ �X�N���[���o�[���g���āADNA�z����̓t�H�[����\�������܂��B
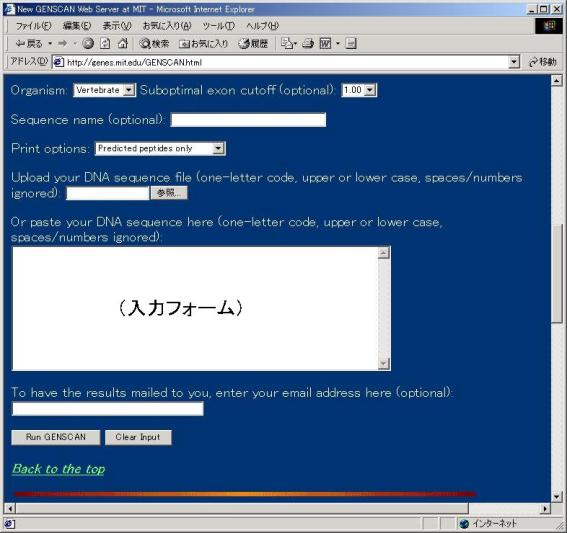
DNA�z����̓t�H�[��
�A �t�H�[����DNA�z�����͂��܂��i�R�s�[���y�[�X�g�œ��͂��܂��j�B
�����ł́A�uHomo sapiens endothelial
nitric oxide synthase�i�q�g��_�����f�����y�f�AACCESSION
ID: D26607�j�v��DNA����z����g���Ď����Ă݂܂��B�Q�m���l�b�g�i��̎��K���Q�l�j�Ȃǂ̃f�[�^�x�[�X�Ō������Ă��������B
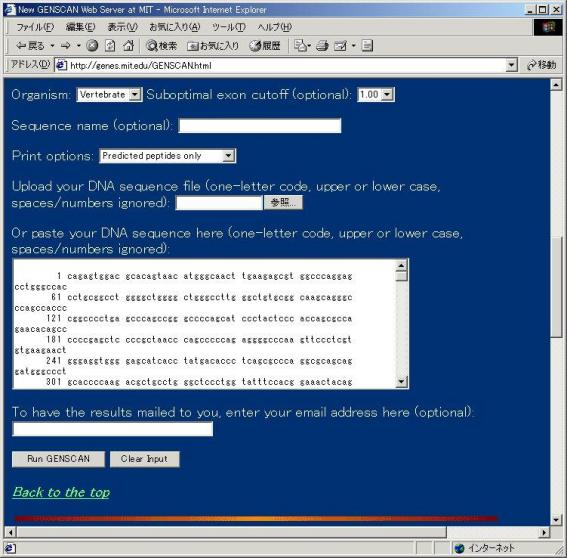
DNA�z���͂��ꂽ�t�H�[��
|
�Q�D�R �e�I�v�V�����̐ݒ� |
��
GENSCAN�̎��s�I�v�V������ݒ肵�܂��B
���̓t�H�[���̊e�I�v�V����
���e�I�v�V�����̐���
|
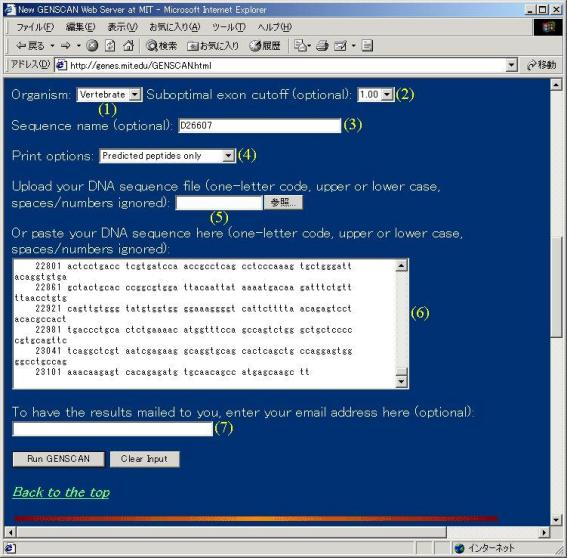
(1) Organism: |
���͂���DNA�z��̐������I�����܂��B ��
Vertebrate�i�Ғœ����j ��
Maize�i�g�E�����R�V�j �� Arabidopsis�i�V���C�k�i�Y�i�j |
|
(2) Suboptimal exon cutoff (optional): |
�i�ȗ��j�����̉��ق����ꍇ�A���̒l��Ⴍ�ݒ肵�܂��B |
|
(3) Sequence name (optional): |
�i�ȗ��j�V�[�N�G���X�̖��O����͂��܂��B |
|
(4) Print options: |
��
Predicted peptides only �\�����ꂽ��`�q�̃A�~�m�_�z���\�������܂��B ��
Predicted CDS and
peptides �\�����ꂽ��`�q�̉���z��ƃA�~�m�_�z���\�������܂��B |
|
(5) Upload your DNA sequence file
(one-letter code, upper or lower case, spaces/numbers ignored): |
�i�ȗ��j�����̃p�\�R����ɂ���DNA�z��̃t�@�C�����w�肷�邱�Ƃ��ł��܂��B |
|
(6) Or paste your DNA sequence here
(one-letter code, upper or lower case, spaces/numbers ignored): |
���̓t�H�[����DNA�z�����͂��܂��BDNA�z���1�����\�L�iA,T,G,C�j�ł���K�v������܂��B�܂��A�啶���Ə������̋�ʂ͂Ȃ��A�z�ɃX�y�[�X�␔�����}������Ă��Ă���������܂��B |
|
(7) To have the results mailed to you,
enter your email address here (optional): |
�i�ȗ��j�t�H�[����E���[���A�h���X����͂���ƁA���ʂ�E���[���ő��M����܂��B |
�@ ����́A�Ώۂ��q�g�̍y�f�ł��邽�߁A
�E
(1) Organism �� Vertebrate�i�Ғœ����j
�̃I�v�V������I�����A
�E
(3) Sequence name �� D26607�iACCESSION ID�j
��⑫���Ƃ��ē��͂��܂��傤�B
|
�Q�D�S ���s�ƌ��ʂ̕\�� |
�� ���s�{�^���������Ď��s���܂��B
���s�{�^��

|
�R�D�P ��`�q�̗\������ |
��
GENSCAN���s��ɏo�͂���錋�ʂ��ꍀ�ڂ����Ă݂܂��傤�B
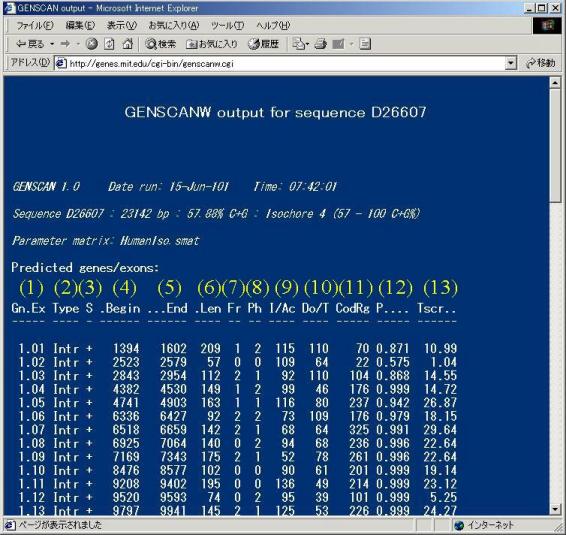
�\�����ꂽ�G�L�\��
���e���ڂ̐���
|
(1) Gn.Ex |
��`�q�̎��ʔԍ� |
|
(2) Type |
��
Init = Initial exon �J�n�R�h���ƃh�i�[���ʂŋ��܂��G�L�\���B ��
Intr = Internal exon �A�N�Z�v�^�[���ʂƃh�i�[���ʂŋ��܂��G�L�\���B ��
Term = Terminal exon �A�N�Z�v�^�[���ʂƃX�g�b�v�R�h���ŋ��܂��G�L�\���B ��
Single-exon gene �C���g�����̂Ȃ���`�q�B�iInit +
Intr + Term = Single-exon gene�j ��
Prom = Promoter �v�����[�^�[�B�]�ʂ��n�܂�DNA��̗̈�B ��
PlyA = poly-A signal �|��A�z��B�^�j������mRNA3�f���[�ɕ��ՓI�ɑ��݂���z��B |
|
(3) S |
���̑� |
|
(4) Begin |
�G�L�\���̊J�n�ʒu |
|
(5) End |
�G�L�\���̏I���ʒu |
|
(6) Len |
�G�L�\���̒��� |
|
(7) Fr |
�|�̓ǂݘg |
|
(8) Ph |
�ǂݘg�̂����i�G�L�\������3�Ŋ������]��j |
|
(9) I/Ac |
3�e���[�����E�̓��_ |
|
(10) Do/T |
5�e���[�����E�̓��_ |
|
(11) CodRg |
�G�L�\���̓��_ |
|
(12) P�c. |
�G�L�\���̊m�� |
|
(13) Tscr.. |
�G�L�\���P�Ƃ̓��_�iLen, I/Ac, Do/T, CodRg�̒l�ŎZ�o����܂��j |
�\�����ʂ���
��
Gn.Ex 1.01�`1.27��27���̃G�L�\�����\������Ă��܂��B
��
Type������\�����ꂽ�G�L�\���̎�ނʂ��܂��B
����̌��ʂł͊J�n�R�h���͊܂܂�Ă��炸�A26�f�Ђ̃G�L�\���ƂP�f�Ђ̏I�~�R�h�����܂ރG�L�\�����\������Ă��܂��B
��
�G�L�\���̑��݊m���͌����ݍ������̂ł����AGn.Ex 1.02��1.26�̗\���G�L�\���ł͊m����0.5��Ƒ��̗\���G�L�\�������Ⴍ�Ȃ��Ă��܂��B
|
�R�D�Q �}�̕\�� |
��
���ʂ��킩��₷���}�Ō��邱�Ƃ��ł��܂��B
��
PDF�t�@�C��
��
PostScript�t�@�C��
��2��ނ�����܂��B�����ł�PDF�t�@�C����\�����Ă݂܂��傤�B
�iPDF�t�@�C���̕\���ɂ�Adobe Acrobat Reader���K�v�ł��B�j
�@ PDF�t�@�C���ւ̃����N���N���b�N����PDF�t�@�C����\������B
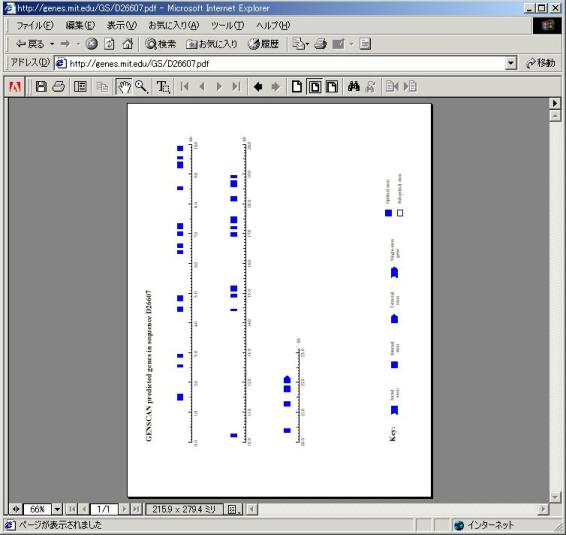
�o�͂��ꂽPDF�t�@�C��
�� Key�ꗗ
|
PDF�t�@�C�����̕\�� |
Key: |
Key�̈Ӗ� |
|
|
Initial exon |
�J�n�R�h���ƃh�i�[���ʂŋ��܂��G�L�\�� |
|
|
Internal exon |
�A�N�Z�v�^�[���ʂƃh�i�[���ʂŋ��܂��G�L�\�� |
|
|
Terminal exon |
�A�N�Z�v�^�[���ʂƃX�g�b�v�R�h���ŋ��܂��G�L�\�� |
|
|
Single-exon gene |
�C���g�����̂Ȃ���`�q |
|
|
Optional exon |
�G�L�\���̏���� �I�v�V�����Suboptimal exon cutoff���Ⴍ�ݒ肷��ƕ\�������B |
|
|
Suboptional exon |
�G�L�\���̏��X��� �I�v�V�����Suboptimal exon cutoff���Ⴍ�ݒ肷��ƕ\�������B |
�R�D�P�̌��ʂƏƂ炵���킹�Č��Ă݂܂��傤�B
��
27�{�̃G�L�\�������l�p�ŕ\������Ă��܂��B
��
���[�i22.0kb�t�߁j�̃G�L�\��������Terminal exon�Ƃ��ăz�[���x�[�X�^�̐}�ŕ\������Ă��܂��B
|
�R�D�R �A�~�m�_�z��̏o�� |
��
�\�����ꂽ�G�L�\����|���A�~�m�_�z�o�͂���܂��B

|
�Q�l���� |
�E
Burge, C. and Karlin, S. (1997) Prediction of
complete gene structures in human genomic DNA. J. Mol. Biol. 268, 78-94.
�E
Burge, C. B. (1998) Modeling dependencies in
pre-mRNA splicing signals. In Salzberg, S., Searls, D. and Kasif, S., eds.
Computational Methods in Molecular Biology, Elsevier Science, Amsterdam, pp.
127-163.
�E
Burge, C. B. and Karlin, S. (1998) Finding
the genes in genomic DNA. Curr. Opin. Struct. Biol. 8, 346-354.
�E
The New GENSCAN Web Server at MIT�ihttp://genes.mit.edu/GENSCAN.html�j
�E
��g �����w���T ��4��